La PCR (Polymerase Chain Reaction = Réaction de polymérisation en chaine) est une technique de biologie moléculaire qui permet d'amplifier un fragment précis d'ADN.
La technique a été inventée par Kary Mullis en 1985 (Prix Nobel de chimie en 1993), et se développe dès la fin des années 1980 grâce à la commercialisation d'une ADN polymérase résistante aux températures élevées (la Taq polymérase), qui permet une automatisation de la technique.
Principe de la PCR⚓
Une petite vidéo très simple qui présente le principe de la PCR
Principe⚓
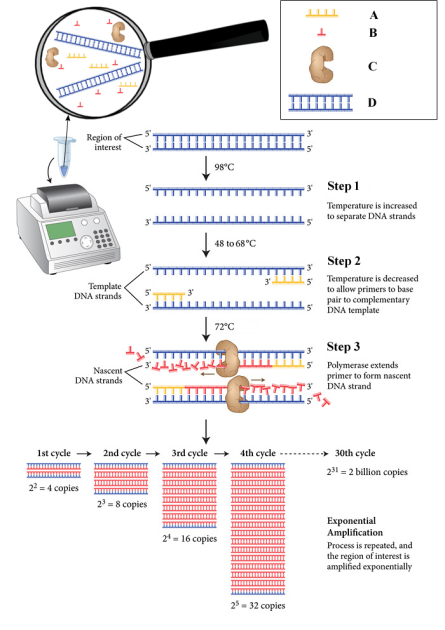
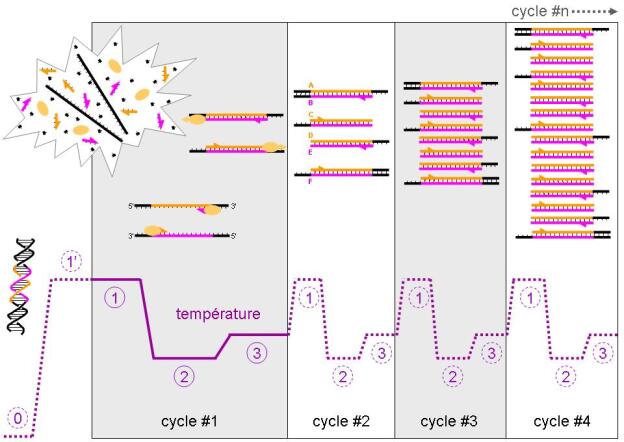
La PCR permet l'amplification d'un fragment d'ADN par une ADN polymérase thermorésistante à partir d'amorces encadrant la région à amplifier. Ces amorces sont complémentaires à une séquence du brin d'ADN d'origine.
Les brins synthétisés à chaque cycle servent de matrice pour le cycle suivant.
Les étapes de la PCR⚓
Introduction⚓
Etapes de la PCR⚓
Chaque cycle de PCR se déroule en 3 étapes à trois températures différentes :
la dénaturation à 95°C qui détruit les liaisons faibles permettant la cohésion des 2 brins d'ADN. Cette étape permet donc de séparer les brins d'ADN pour obtenir de l'ADN simple brin sur lequel les amorces pourront se fixer.
l'hybridation des amorces sur l'ADN simple brin. La température de cette étape dépend de la nature des amorces
l'élongation (ou extension), c'est-à-dire l'addition successive à l'extrémité 5' de l'amorce des désoxynucléotides complémentaires à l'ADN matrice par l'ADN polymérase. Cette étape se fait à 72°C, température optimale d'activité de la Taq polymérase.
L'amplification⚓
A chaque cycle, on double la quantité d'ADN synthétisé. Ainsi, à l'issue des 30 cycles traditionnellement réalisés, on a multiplié par 230 (≈109) le nombre copies de la séquence cible. On obtient alors une quantité d'ADN suffisante pour être détectée sur gel d'agarose.
Les composants de la PCR⚓
L'ADN polymérase⚓
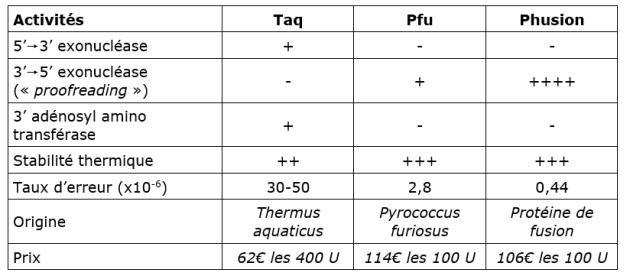
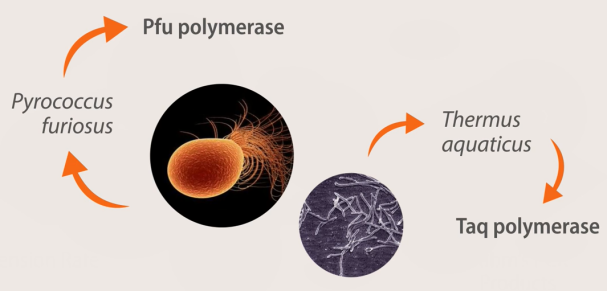
Les ADN polymérases utilisées aujourd'hui en PCR sont extraites de bactéries vivant naturellement à des températures élevées.
La plus utilisée est l'ADN polymérase Taq extraite de Thermus aquaticus, mais il en existe d'autres comme la polymérase Pfu extraite de Pyrococcus furiosus, une archée hyperthermophile (= se développe entre 70 et 103°C).
Voyons à partir de cette vidéo les propriétés d'une polymérase adaptée à la PCR.
Caractéristique des ADN polymérases utilisées en PCR⚓
Le choix de la polymérase dépend de :
sa thermostabilité,
sa processivité, c'est-à-dire la probabilité qu'elle se détache de l'ADN pendant l'extension (important pour des amplicons de grande taille).
sa vitesse d'élongation (la Taq allonge à la vitesse de 1 kb par minute mais les enzymes de nouvelle génération vont généralement 4 fois plus vite)
sa fidélité ; certaines polymérases possèdent une activité exonucléase 3'-5' (proofreading) qui permet la correction des erreurs (mais élimine aussi le A en 5' ce qui les rend inadaptées pour le TA-cloning - voir TP B2BTK)
Les polymérases Hot-Start⚓
Le manque de spécificité d'une PCR est lié à une hybridation non spécifique des amorces. Cette hybridation non spécifique est d'autant plus important que la température est basse (l'agitation thermique est moindre donc les interactions d'affinité modérée ne sont pas rompues).
Il existe donc un risque important AVANT le début de la PCR, alors que les tubes restent à température ambiante.
Pour y remédier, les laboratoires ont développé des polymérases dites "Hot-start", inactives avant la première étape de la PCR.
Pour comprendre le principe d'une PCR "Hot start"⚓
Les ADN polymérases "hot start" sont inactives avant la première étape de la PCR (chauffage à 95°C pendant environ 5 min). Comment est-ce possible ?
Température et protéine
Température et inhibiteur de la polymérase thermostable
Principe de la PCR hot-start⚓
Une polymérase "hot-start" est associée à un inhibiteur protéique (anticorps) ou nucléotidique (aptamère). Lors de la première étape de dénaturation à 95°C de la PCR, cet inhibiteur sera dénaturé et se détachera de la polymérase qui pourra alors effectuer la PCR normalement.
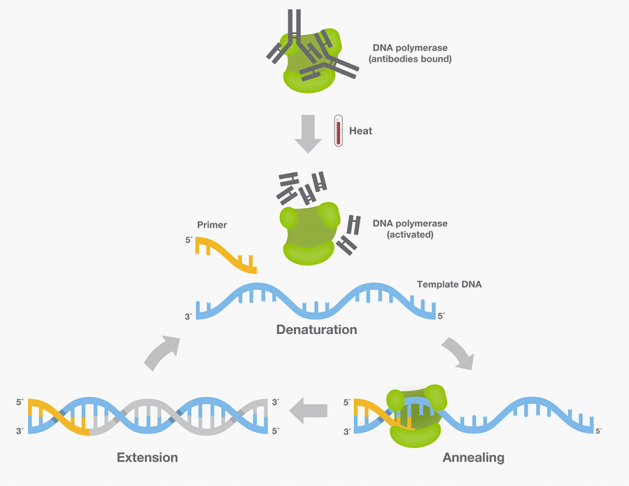
Remarque : il existe des variantes qui agissent non pas sur la polymérase mais sur la composition du tampon, libérant le Mg2+ nécessaire à la polymérase à des températures supérieures à 70°C.
L'ADN cible⚓
La PCR peut permettre de révéler des quantités très faibles d'ADN mais il faut que la séquence cible soit intacte (taille généralement entre 0,1 et 3 kb). On utilise en général 5 à 500ng d'ADN.
Voyons un peu plus en détail les contraintes liées à l'ADN cible à l'aide de cette vidéo
Les amorces (= primers)⚓
Rappel : brin codant et non codant⚓
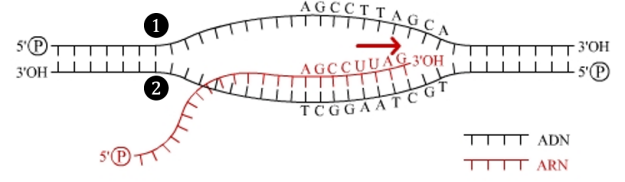
Identification des brins
Emplacement des amorces⚓
Les amorces doivent encadrer la région à amplifier et s'hybrider chacune sur un brin différent.
L'une des amorces est complémentaire au brin codant (donc même séquence que le brin non codant ou antisens) ; on l'appelle amorce antisens (ou Reverse en anglais)
L'autre amorce est complémentaire au brin non codant (donc même séquence que le brin codant ou sens) ; on l'appelle amorce sens (ou Forward en anglais)
Pour s'entraîner...⚓
Soit un fragment d'ADN de 12 kb pour lequel on dispose de 10 amorces.

Orientation des amorces
Amplification ?
Détermination de la séquence des amorces⚓
La détermination de la séquence des amorces est effectuée par une approche bio-informatique en utilisant des banques de séquences (voir cours de bio-informatique de B2BTK)
Caractéristiques des amorces⚓
D'une manière générale, le choix d'un couple d'amorces tient compte des paramètres suivants :
en général 18 à 24 nucléotides pour avoir une spécificité suffisante tout en conservant une hybridation efficace
températures d'hybridation des 2 amorces proches et inférieures à la température d'élongation. Dans l'idéal, la température d'hybridation devrait se situer entre 50 et 67°C.
absence de séquences inversées répétées qui conduiraient à la formation de structures secondaires à l'intérieur de l'amorce
absence de complémentarité entre les 2 amorces pour empêcher l'hybridation entre amorces
Température d'hybridation⚓
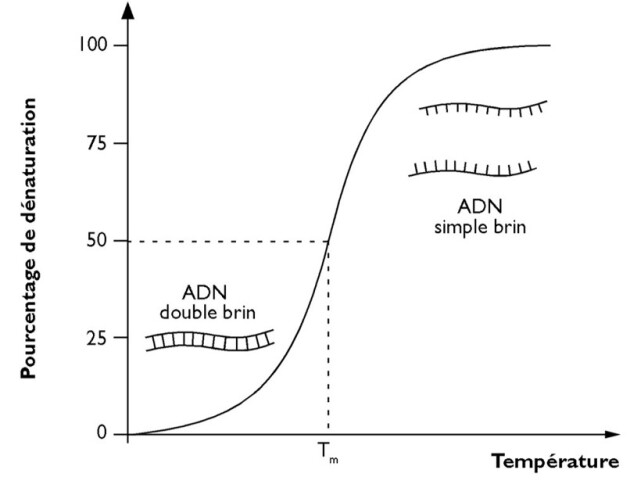
Le choix de la température d'hybridation dépend de la longueur et de la composition des amorces. Classiquement, on se place 5 à 10°C au-dessous de la température de fusion.
La température de fusion Tm (= melting temperature = 50% ADN dénaturé) est donnée par la formule de Wallace (pour des amorces de moins de 30 nucléotides) : Tm = 2(A+T) + 4(G+C).
Note : les bases G et C ont plus de poids car les paires G-C sont associées par 3 liaisons hydrogène et sont donc plus difficiles à dissocier que les paires A-T qui n'en comptent que 2.
Si les 2 amorces n'ont pas la même température d'hybridation, on essaie de se placer à une température qui pourrait convenir aux deux. Sinon, on se base sur la Tm la plus faible.
Pour s'entraîner...⚓
On souhaite amplifier par PCR le fragment du gène de l'alcool déshydrogénase (ADH) de S.cerevisiae compris entre les résidus 908 et 1078 à l'aide d'amorces de 21 paires de bases. La séquence codante est indiquée ci-dessous :

Sauf indication contraire, une séquence est toujours donnée dans le sens 5'-3' et il s'agit de la séquence codante.
Taille de l'amplicon
Séquence des amorces
Température d'hybridation
Les désoxyribonucléotides triphosphates⚓
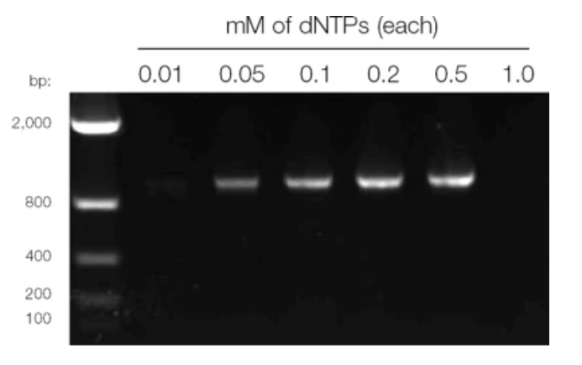
Des désoxyribonucléoside triphosphates libres (dNTP) en excès sont nécessaires pour la synthèse de l'ADN. Les quatre dNTP doivent être utilisés à des concentrations équivalentes pour limiter les erreurs d'incorporation.
Influence de la concentration en dNTP
Le tampon de réaction⚓
Le tampon de réaction est généralement fourni à la concentration 10X et sa composition varie selon la polymérase utilisée.
Voyons à partir de cette vidéo comment le tampon peut influer sur la qualité de la PCR.
La PCR en pratique⚓
Et si on faisait notre petit TP virtuel ?!...
Le but de ce TP est de mettre en évidence l'influence de la concentration en ions magnésium sur l'amplification d'une séquence du pBR322 par la Taq polymérase et de déterminer la concentration optimale requise pour la réaction.
Pour cela, on réalisera une amplification par PCR d'une séquence du plasmide pBR322 à partir d'une colonie d'E.coli transformée par pBR322. On utilisera différents volumes de magnésium et on vérifiera la qualité de la PCR par électrophorèse sur gel d'agarose
Un extrait de la séquence du plasmide pBR322 est donné ci-dessous :

Les contraintes de la PCR⚓
Il est indispensable non seulement d'éviter toute contamination par un ADN exogène qui pourrait servir de matrice et fausser les résultats, mais aussi d'apporter des DNases exogènes qui pourraient endommager l'échantillon d'ADN.
Pour cette raison,
les manipulations s'effectuent avec des gants neufs,
on utilise de l'eau de qualité « Biologie Moléculaire »,
on utilise du matériel stérile à usage unique (microtubes de 0,2 ml, cônes)
Préparation du mélange réactionnel⚓
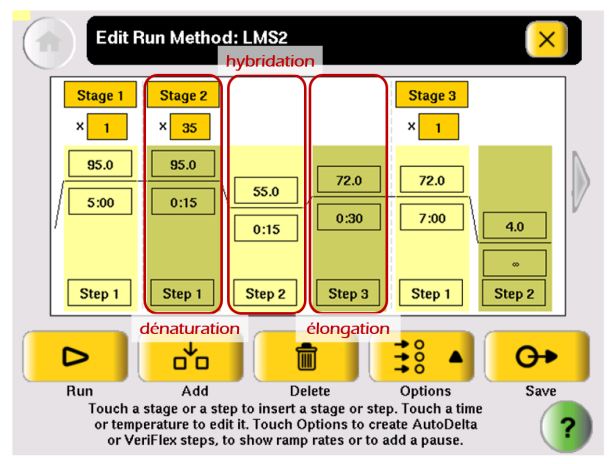
Programme d'amplification⚓
Le programme d'amplification est à adapter à chaque PCR. Il comporte néanmoins les phases suivantes :
Dénaturation initiale : 95°C pendant 3 min Cette étape permet, outre la séparation des 2 brins d'ADN, de casser les structures secondaires et de dénaturer d'autres enzymes qui pourraient se trouver dans la solution
30 cycles d'élongation :
dénaturation : 95°C pendant 30 secondes
hybridation : à la THyb appropriée pendant 45 secondes
polymérisation : 72 °C (= T° optimale de la Taq) pendant 1 minute
Extension finale (extension des brins inachevés) : 72°C pendant 5 min
A l'issue de la PCR, le thermocycleur maintient les tubes à +4°C
Température d'hybridation
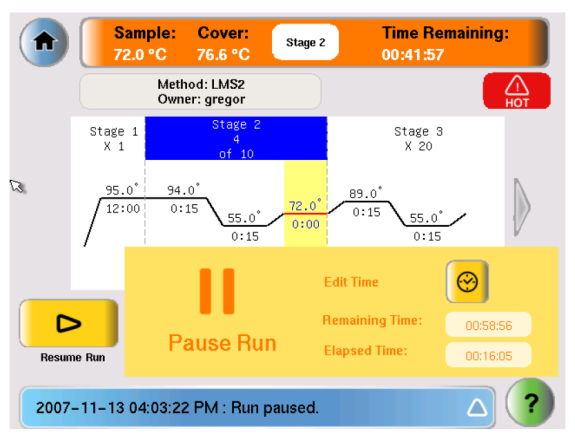
Mais au fait, à quoi ressemble un thermocycleur ?⚓
Un thermocycleur a la taille d'une grosse friteuse. Il possède un écran de contrôle pour régler les paramètres d'amplification et suivre la progression des cycles.

Il permet de régler le nombre, la durée et la température des cycles.
Les tubes sont ensuites placés à l'intérieur du thermocycleur et la PCR peut démarrer.
Une fois la PCR en route, on peut suivre le déroulement des phases sur l'écran.
Électrophorèse des produits de PCR⚓
On réalise ensuite une électrophorèse des produits de PCR sur gel d'agarose 1%
Préparation des échantillons⚓
Taille attendue des amplicons⚓
Position des amorces
Question⚓
Repérer la position des amorces sur la séquence partielle du pBR322 en indiquant le sens de lecture de la polymérase à partir de chaque amorce.
Solution⚓
L'amorce forward (sens) a la même séquence que le brin codant ; elle est complémentaire au brin non codant au niveau de sa séquence (5' CTCGCTCTGCTAATCCTGT 3') ; elle est lue directement sur le brin codant. Au contraire, l'amorce reverse (antisens) est complémentaire au brin codant (voir séquence)
La Taq polymérase polymérise l'ADN dans le sens 5'-3' (= à partir d'une extrémité 3'OH libre)
Taille de l'amplicon
Analyse du gel⚓
Les résultats obtenus pour un groupe de TP sont les suivants :
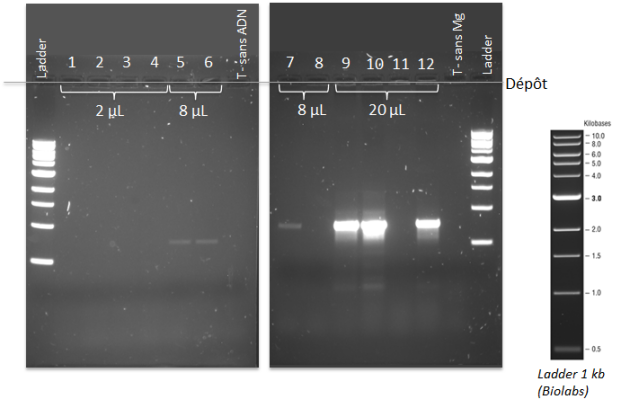
Rôle du témoin
Analyse en fonction du volume de MgCl2
Conclusion
Explication
Une variante de la PCR, la PCR quantitative (= PCR en temps réel)⚓
La PCR quantitative est une variante de la PCR couramment utilisée en laboratoire, aussi bien en recherche qu'en diagnostic.
Découvrons-la en vidéo
Principe⚓
La PCR en temps réel ou PCR quantitative (qPCR) permet de quantifier la quantité (nombre de copies) d'ADN initialement présente dans le mélange réactionnel en détectant à chaque cycle la fluorescence émise par les amplicons néoformés.
En utilisant un marqueur qui fluoresce uniquement lorsqu'il y a amplification, on peut repérer à partir de quel cycle la fluorescence est détectable.
En comparant ce seuil à une courbe étalon, on peut en déduire le nombre de copies initialement présentes dans le mélange (plus il y a de copies initiales, plus la détection sera rapide).
Applications⚓
La PCR en temps réel est utilisée
en diagnostic pour calculer la charge virale (en coronavirus par exemple)
ou en recherche pour étudier le niveau d'expression d'un gène.
L'expression d'un gène étant directement corrélée au nombre de molécules d'ARNm dans le cytoplasme de la cellule, on passe au préalable par une phase de transcription inverse afin de convertir l'ARNm en ADN complémentaire
Courbe d'amplification⚓
La courbe d'amplification représente la cinétique de la réaction. On distingue 3 phases :
Phase d'initiation = bien que le nombre de produits de PCR augmente de manière exponentielle, la fluorescence émise ne se distingue pas de la ligne de base
Phase exponentielle = visualisation du doublement de la quantité d'ADN à chaque cycle
Plateau = épuisement des réactifs + inhibition de la polymérase par le nombre élevé d'amplicons qui se renaturent entre eux plutôt que s'hybrider aux amorces
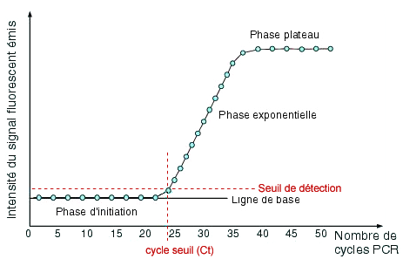
On appelle cycle seuil (Ct = threshold cycle ou Cq = quantification cycle), le cycle à partir duquel la fluorescence devient significative. Le Ct dépend directement du nombre d'amplicons dans le milieu réactionnel.
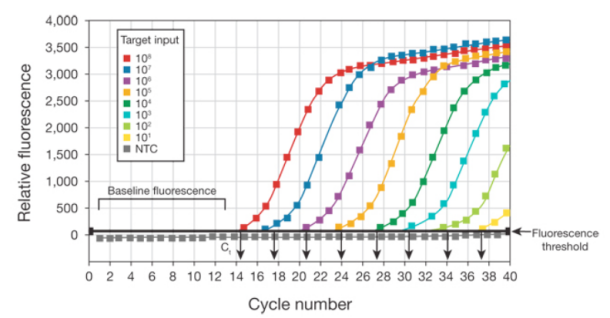
Puisque le nombre d'amplicons double à chaque cycle, le seuil sera atteint d'autant plus rapidement que le nombre de copies dans l'échantillon d'origine est élevé. Un Ct faible correspond donc à un nombre de copies initiales élevé.
A partir d'échantillons de référence, on peut ainsi tracer une droite d'étalonnage pour quantifier le nombre de séquences initiales dans les échantillons à analyser.
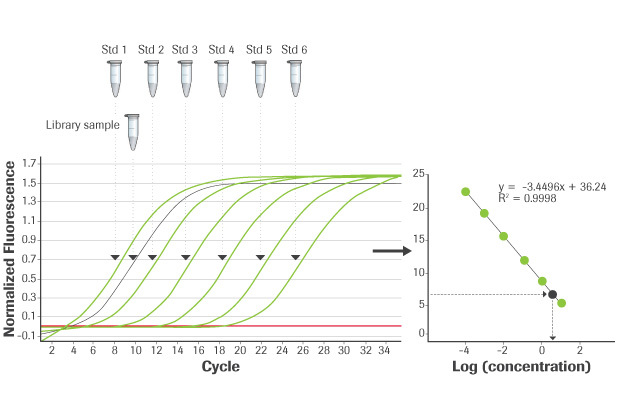
Remarque : plus il y a de copies dans le milieu réactionnel initial, plus la fluorescence sera rapidement détectable et donc plus Ct sera faible ; on aura donc une droite d'étalonnage décroissante, inversement proportionnelle au log du nombre de copies.
Méthodes de détection⚓
Plusieurs méthodes sont utilisées pour quantifier l'ADN. Voyons les plus connues.
Détection au SYBR Green⚓
Principe⚓
Le SYBR Green fluoresce lorsqu'il est associé à l'ADN double brin. Durant la phase d'élongation, on aura donc une augmentation de la fluorescence avec l'allongement des amplicons. On mesure donc la fluorescence à la fin de chaque cycle d'élongation, là où elle est maximale.
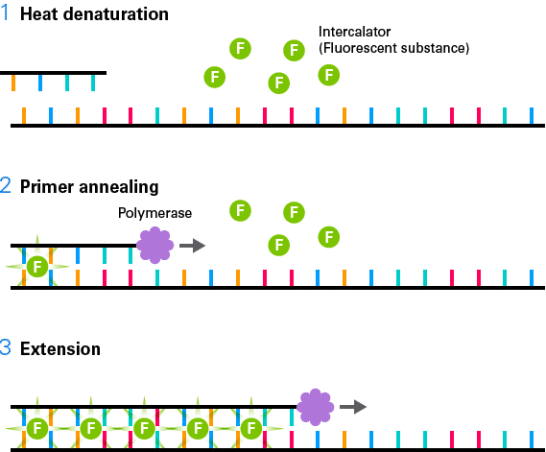
La fluorescence est proportionnelle au nombre d'amplicons.
Les limites de la méthode⚓
Le SYBR Green s'associant à n'importe quel ADN double brin, il ne permet pas de distinguer si la fluorescence mesurée correspond bien à l'amplicon souhaité ou à un éventuel contaminant.
Une solution⚓
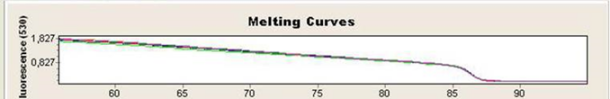
Afin de s'assurer de la qualité de l'amplification, on réalise une courbe de fusion en mesurant la fluorescence émise en fonction de la température.
La température de fusion dépend de (voir cours propriétés de l'ADN) :
la longueur du fragment d'ADN
la richesse en GC
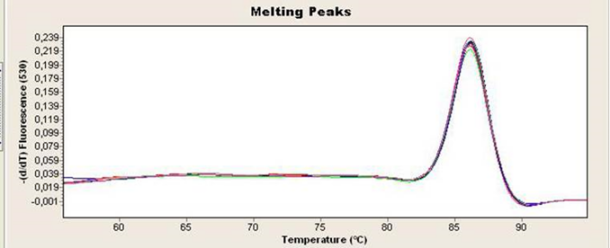
Elle est donc caractéristique d'une séquence donnée. Si tous les échantillons ont la même Tm, c'est qu'il s'agit de la même séquence donc qu'on a bien un même amplicon (donc un seul ADN amplifié).
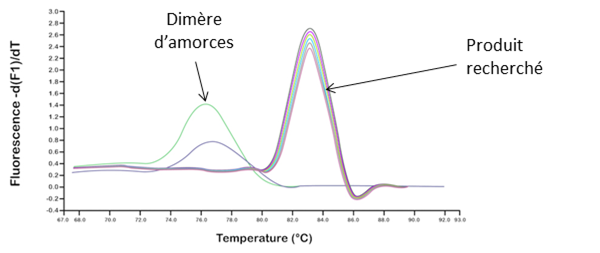
Pour réaliser une courbe de fusion, on fait varier la température de 95°C à la température d'hybridation pour que l'ADN se ré-apparie correctement puis de la température d'hybridation à 95°C et on enregistre la fluorescence émise ; celle-ci est proportionnelle à la quantité d'ADN double brin. Elle va donc diminuer lorsque l'ADN va passer de l'état double brin à simple brin.
On fait ensuite la dérivée de cette courbe (fait ressortir les variations de pente). C'est cette dérivée qu'on appelle courbe de fusion dans le cas de la qPCR.
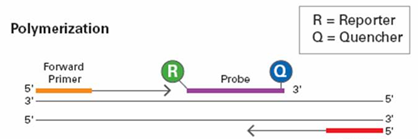
Détection par hydrolyse d'une sonde (Taqman)⚓
Les sondes de type TaqMan sont des sondes d'hydrolyse qui permettent d'accroitre la spécificité de la qPCR.
On utilise une sonde spécifique de la région à amplifier. Un fluorophore rapporteur y est fixé à l'extrémité 5' et un fluorochrome suppresseur (quencher) à l'extrémité 3'. Le suppresseur absorbe la fluorescence émise par le fluorophore rapporteur lorsqu'il se trouve à proximité de celui-ci (FRET), donc tant que la sonde est intacte.
La Taq polymérase possède une activité exonucléase 5'-3'. Pendant l'élongation, alors que la Taq allonge l'amorce, son activité exonucléase va dégrader la sonde hybridée au brin matrice, relarguant le fluorophore rapporteur qui s'éloigne du suppresseur dont la fluorescence va alors pouvoir être détectée.
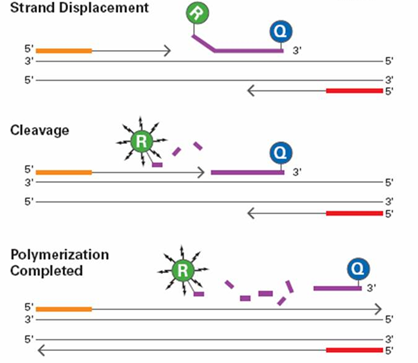
La fluorescence émise est proportionnelle au relargage du fluorophore donc à la quantité d'amplicon (1 sonde par amplicon). On est ici certain que la fluorescence émise correspond à l'amplification de la sonde d'intérêt.
L'extinction de fluorescence fait intervenir 2 fluorophores proches : une donneur et un accepteur.
On excite le donneur par sa longueur d'onde d'excitation λex
La fluorescence émise par le donneur va exciter l'accepteur qui peut émettre une fluorescence ou neutraliser la fluorescence du donneur.
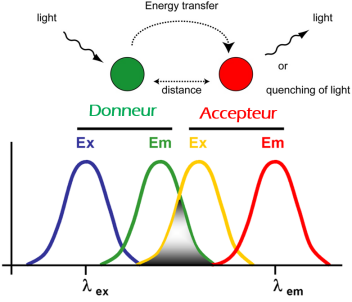
Pour s'entraîner...⚓
Détection de la fluorescence par hybridation de 2 sonde (HybProbe)⚓
Dans cette méthode, on utilise 2 sondes spécifiques de la région à amplifier : l'une marquée avec un fluorophore donneur à l'extrémité 3' et l'autre avec un fluorophore accepteur en 5'. Quand les 2 fluorophores sont proches (moins de 10 nt d'écart), la lumière émise par le fluorophore donneur va exciter le fluorophore accepteur. On parle de transfert d'énergie entre molécules fluorescentes (FRET = Fluorescence Energy Transfert).
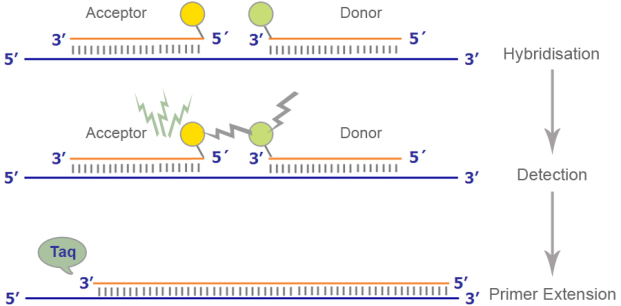
Lorsque les 2 sondes sont éloignées (en solution), on n'observe qu'un bruit de fond fluorescent. Pendant l'étape d'hybridation, les 2 sondes se fixent sur leurs séquences cibles respectives de la région à amplifier (localisées à moins de 10 nt d'écart). La proximité permet le FRET et la fluorescence du fluorophore accepteur peut être détectée. Au cours de l'élongation, la Taq polymérase déplace les sondes qui retournent en solution dans le milieu.
Un petit coup de blues ?...⚓
de l'humour scientifique pour se remonter le moral
(Le confinement commence à faire des ravages !)