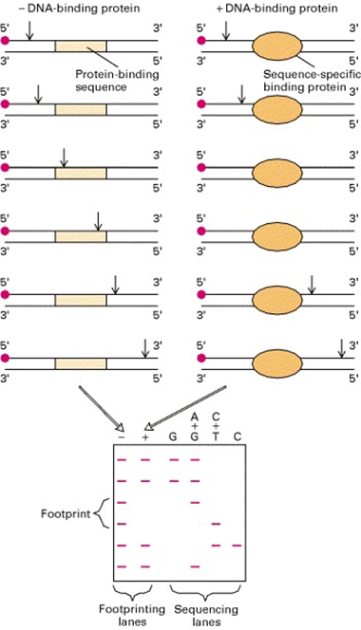
On a essayé de mettre en évidence les séquences auxquelles l'ARN polymérase se fixe par une expérience dite de « DNA footprinting » (empreinte ADN).
Principe de l'expérience⚓
Un fragment d'ADN est marqué par 32P en 5' puis incubé en présence ou non d'ARN polymérase. Il est ensuite soumis à une digestion partielle par la DNase I, une endonucléase qui hydrolyse les liaisons phosphodiesters de manière aléatoire. On utilise une faible concentration de DNase I de manière à ce qu'elle clive une seule fois chaque brin.
Les fragments sont ensuite séparés des protéines, dénaturés et analysés sur gel de polyacrylamide par autoradiographie. Les résultats sont présentés dans les 2 premières pistes du document ci-dessous :
Rappel :
Rappel : sur gel de polyacrylamide 2 bandes consécutives ont une taille qui diffère de 1 nucléotide.
Empreinte à la DNase I⚓
On analyse d'abord les 2 première pistes du gel.
Identification sur gel
Piste (-)
Piste (+)
Interprétation des différences
Séquençage⚓
On renouvelle l'expérience en clivant cette fois à l'aide d'agents chimiques qui clivent après un nucléotide défini selon la méthode de séquençage de Maxam et Gilbert.
Les résultats correspondent aux 4 puits suivants.
Complément :
Les résultats présentés dans le document correspondent à une expérience-type et non spécifiquement à l'ARN polymérase ; la séquence identifiée ne correspond donc pas en réalité à la région promotrice.
Comprendre le gel⚓
Le séquençage par la méthode de Maxam et Gilbert est basé sur le clivage chimique après certaines bases. On utilise donc traitements chimiques dans 4 tubes différents et on laisse agir pendant un temps court de telle sorte à avoir au plus 1 coupure par brin.
Statistiquement, on aura donc des coupures après chacune des bases correspondant au réactif. On fait ensuite migrer sur gel de polyacrylamide.
Traitement chimique | Conséquence |
|---|---|
DMS + chauffage | Clivage au niveau des G et A |
DMS + piperidine | Clivage au niveau des G |
Hydrazine + piperidine | Clivage au niveau des C et T |
Hydrazine + NaCl 2M | Clivage au niveau des C |
G
A
T
C
Exploitation des données⚓
On va chercher à identifier la séquence spécifique sur laquelle se fixe l'ARN polymérase (gel).
Séquence ADN
Séquence reconnue par l'ARN polymérase
Séquences reconnues par l'ARN polymérase⚓
On montre que l'holoenzyme se fixe préférentiellement au niveau d'une région située entre les positions -40 et +20 alors que le core peut se lier n'importe où. Si on mute spécifiquement les régions situées en position -35 et -10, alors l'holoenzyme se fixe beaucoup moins bien.
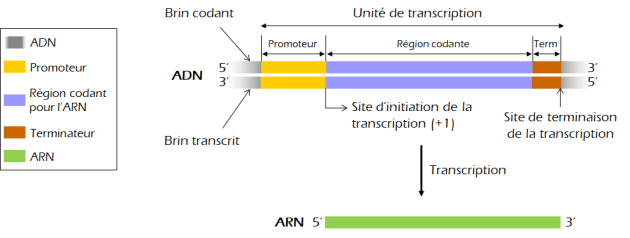
Rappel : Convention d'écriture
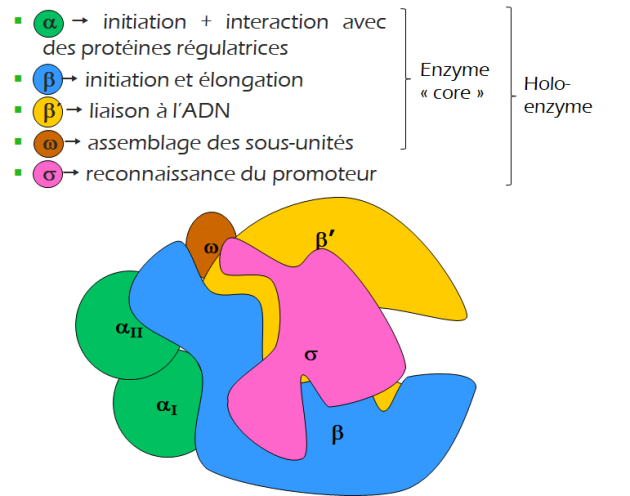
Importance du facteur sigma⚓
Séquences consensus⚓
On appelle séquence consensus, une séquence type d'une région donnée de l'ADN dans laquelle on note la base rencontrée le plus fréquemment à chaque position. Elle est établie par comparaison de séquences réelles.
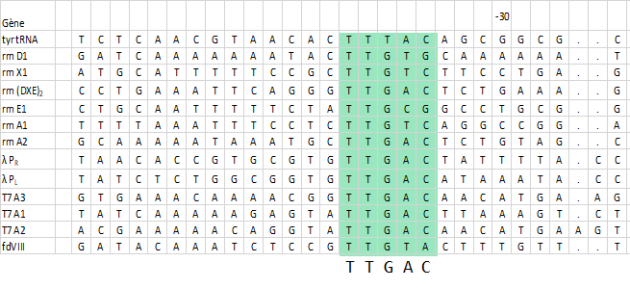
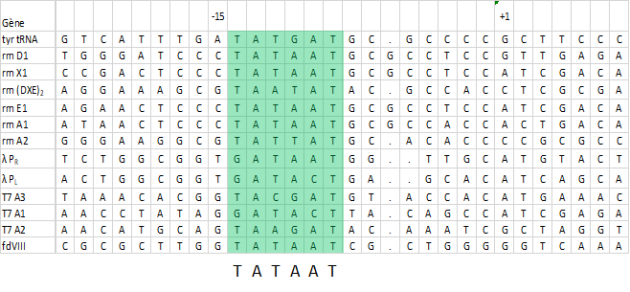
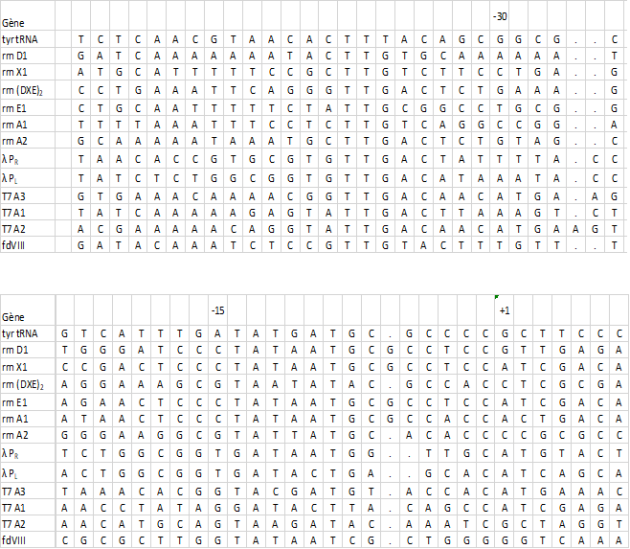
On a séquencé les promoteurs de différents gènes d'E.coli. Leurs séquences (brin codant) sont notées dans le document ci-dessous :
Séquences consensus⚓
Identification des séquences consensus
Particularités de la région -10
Séquence de l'ARN⚓